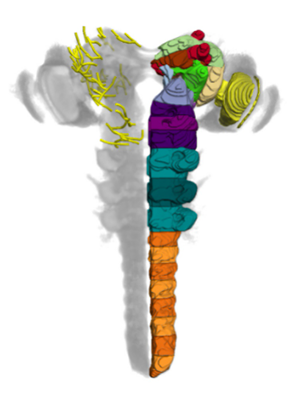
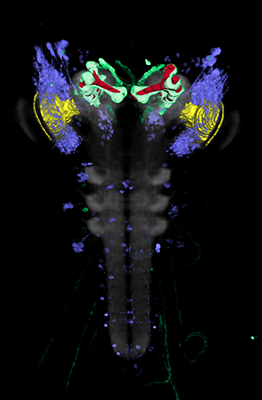
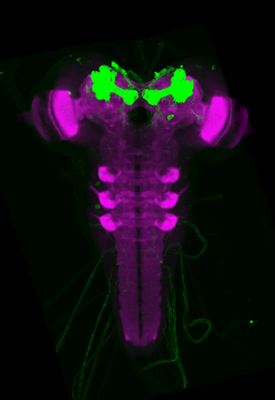
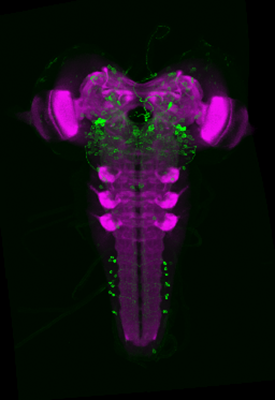
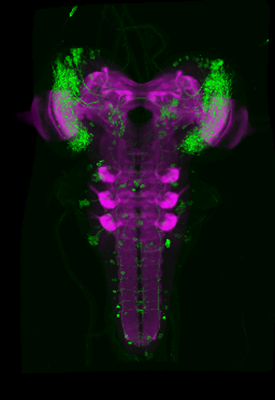
In diesem Projekt erstellen wir erstmals einen dreidimensionalen Gehirnatlas für die Drosophila Larve. Neben einer einheitlichen Nomenklatur integriert dieser Ansatz mehrere tausend genetische Werkzeuge in einer offen zugänglichen Online-Datenbank.
Vorhaben
Wie organisiert ein Gehirn Verhalten basierend auf sensorischem Input, Motivation oder aufgrund erworbener Erfahrung? Seit Jahrhunderten beschäftigt diese Frage Gehirnforscher aus vielen Disziplinen. Obwohl es gelungen ist, bestimmte Aspekte von Verhalten definierten Gehirnarealen zuzuordnen, können interessanterweise diese Ergebnisse nur schwer in Modelle übertragen werden. Daher gibt es auch heute noch kein einfaches, generell akzeptiertes Modell für die Funktionsweise des Gehirns. Das Problem, das sich hierbei stellt ist meist, dass der genaue Schaltplan und die Verenetzung des Gehirns aus vielen tausend Neuronen nicht bekannt ist.
Vorgehen
In diesem Projekt haben wir einen Standardatlas generiert, der es erlaubt vorhandene genetische Werkzeuge zu registrieren. Dadurch sind diese und die darin markierten Zellen in einem kompletten Larvengehirn in 3D darstellbar und über Abfragen suchbar.
Ziel
Es ist das Ziel dieser Arbeit das Gehirn von Drosophila während seiner larvalen Entwicklung in seiner Gesamtheit neuroanatomisch zu rekonstruieren und gleichzeitig tausende von neurogenetischen Werkzeugen (GAL4, LexA, Split-GAL4, ...) zu kartieren, die es erlauben jede einzelne Zelle im Gehirn der Larve genetisch und funktionell zu manipulieren. Dies ist ein wichtiger Schritt, um langfristig zu verstehen wie das einfache Gehirn der Larve funktioniert und dadurch grundlegende neuronale Prinzipien zu erkennen, die man im nächsten Schritt vielleicht auf zellulär gößere Gehirn übertragen kann.
Publikationen
larvalign: Aligning Gene Expression Patterns from the Larval Brain of Drosophila melanogaster
Muenzing SEA, Strauch M, Truman JW, Bühler K, Thum AS, Merhof D.
Neuroinformatics. 2018 Jan;16(1):65-80.
DOI: 10.1007/s12021-017-9349-6